
GFPとDsRedの蛍光比からRNAiの効果を見積もる
トランスフェクション
(1) トランスフェクションを行う24時間前に,24穴プレートに1 wellあたり3×104 個のHeLa細胞をまく(1 wellあたり10 % FBS/DMEM 500 uL)。
(2) 20 uM siRNAを200 nMに希釈する。 20 uM siRNA 0.2 uLをバッファー 19.8 uLに加えてピペッティングする (A)。
(3) Opti−MEM 90 uLに1714 ug/mLのpEGFP 0.36 uL (617 ng),1643 ug/uLのpDsRed (985 ng)、 (A)のsiRNA 0.2 uL (62.6 pg)を加え(※),ピペッティングした後,15分静置 。
(※) siRNAはpEGFPをターゲットにしている ( siRNAを加えればEGFPは発現せず、siRNAを加えないとEGFPは発現する DsRedの発現に影響は無い)
(4) Opti−MEM 9.2 uLにリポフェクトアミン2000 0.8 uLを加え,よくピペッティングした後,10分静置 。
(5) (3)と(4)をピペッティングにより混合し,20分静置
(6) (5) の待ち時間で24穴プレートの培地を交換しておく。 古い培地を除去してからPBS(−)で洗う。PBS(−)を除去してから,1 wellあたり10 % FBS/DMEMを 500 uL加える 。
(7) (6) のプレートに (5) を加え,37 ℃ CO2インキュベーターで24時間カルチャー する。
顕微鏡観察
(8) 用いる蛍光顕微鏡はオリンパスIX-71。対物レンズはX10の〜。観察用光源,ケージ解除用光源(アンケージングする場合),CCDカメラおよびシャッターコントローラーの電源を入れてから,PCの電源を入れて,MetaMorphを立ち上げる。
(9) 細胞の透過像(ターレットを4に合わせる),EGFPの蛍光像(ターレットは2。観察用光源のフィルターを押し込む),DsRedの蛍光像(ターレットは3。観察用光源のフィルターは手前側)を観察し,CCDカメラで画像を取り込む。
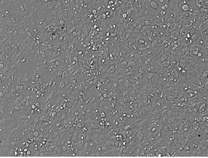 HeLa細胞の微分干渉像 | 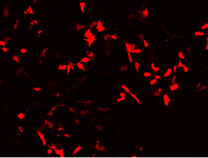 DsRedの蛍光像 |
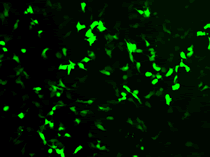 EGFPの蛍光像 |  siRNAでEGFPの発現を抑制した |
データ処理
(10) 取り込んだ画像をEGFP、DsRedともディスプレイ上の左のバーを使って擬似カラーをつけ、auto threshhold light object をクリックし、蛍光の範囲を選択する。
(11) measureのintegrated morphometry analysisをクリックし、setup parameterのmeasuringを選択し、measureをクリックする。
(12) (11) の操作で、蛍光エリアのトータル面積がわかるので、EGFPとDsRedそれぞれの蛍光エリアのトータル面 積を求め、EGFP/DsRedで各々のサンプルの比を求める。
参考文献
お問い合わせ:東邦大学理学部生物分子科学科 古田寿昭
当サイトの画像・文書の転載を一切禁じます。
Copyright© 2004 Furuta lab Toho Univ. All Rights Reserved.